Haplootype studiemetoder, diagnostikk, sykdommer
- 2331
- 331
- Markus Fredriksen
EN Haplootype Det er en region av genomet som har en tendens til å arve sammen gjennom flere generasjoner; Vanligvis er alt lokalisert i samme kromosom. Haplootyper er produktet av genetisk binding og forblir intakt under genetisk rekombinasjon.
Ordet "haplotipo" stammer fra en kombinasjon av ordet "haploid" og ordet "genotype". "Haploid" refererer til celler med et enkelt sett med kromosomer og "genotype" snakker om den genetiske sammensetningen av en organisme.
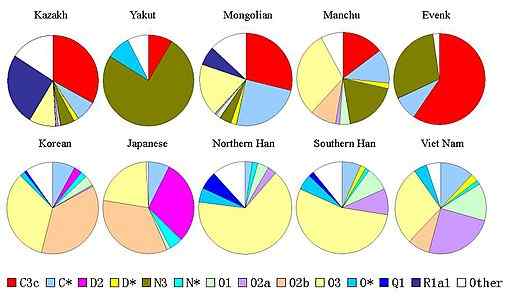
 Skjema for fordelingen av kromosom -haplotyper og i asiatiske populasjoner (kilde: Moogalord [offentlig domene] via Wikimedia Commons) Etter definisjonen kan en haplotype beskrive et par gener eller mer som er arvet sammen på et kromosom fra en foreldre, eller kan beskrive et kromosom som er fullstendig arvet fra en foreldre, som tilfellet er kromosomet og menn.
Skjema for fordelingen av kromosom -haplotyper og i asiatiske populasjoner (kilde: Moogalord [offentlig domene] via Wikimedia Commons) Etter definisjonen kan en haplotype beskrive et par gener eller mer som er arvet sammen på et kromosom fra en foreldre, eller kan beskrive et kromosom som er fullstendig arvet fra en foreldre, som tilfellet er kromosomet og menn.
Når haplotyper for eksempel deler gener for to forskjellige fenotypiske tegn, for eksempel hårfarge og øyenfarge, vil individer som har hårfarge -gen også ha det andre genet for fargen på øynene.
Haplootyper er et av de mest brukte verktøyene i dag for studiet av slektsforskning, for å spore opprinnelsen til sykdommer, for å karakterisere den genetiske variabiliteten og fylogeografien til populasjoner av forskjellige typer levende vesener.
Det er flere verktøy for studiet av halootyper, en av de mest brukte i dag er "Haplotypekart“(HapMap), som er en webside som gjør det mulig å bestemme hva som er genomsegmentene som er haplotyper.
[TOC]
Studiemetoder
Haplootyper representerer en mulighet til å forstå arven etter gener og deres polymorfisme. Med oppdagelsen av teknikken "polymerasekjedereaksjon" (PCR, engelsk "Polymerase kjedereaksjon”) Han avanserte mye i studiet av haplotyper.
Det er for øyeblikket mange metodologier for studiet av haplotyper, noen av de mest høydepunktene er:
DNA -sekvensering og enkelt nukleotidpolymorfismedeteksjon (SNP)
Utviklingen av nye generasjons sekvenseringsteknologier representerte et stort sprang for studiet av haplotyper. Nye teknologier tillater variasjoner av opptil en enkelt nukleotidbase i spesifikke regioner av en haplotype.
Kan tjene deg: Rekombinant DNA: Teknikk, applikasjoner og fundamenterI bioinformatikk brukes også begrepet haplotype for å referere til arven av en enkelt nukleotidpolymorfismer (SNP) i DNA -sekvenser.
Å kombinere bioinformatikkprogrammer med påvisning av haplotyper ved bruk av ny generasjons sekvensering kan nøyaktig identifiseres posisjon, erstatning og effekt av endring av hver base på genomet til en populasjon.
Microsatellites (SSRS)
Mikrosatellittene eller SSR -ene henter navnet sitt fra engelskmennene “Simplementere gjentakelse og Kort tandem gjenta"". Dette er korte nukleotidsekvenser som gjentas suksessivt innenfor et genomregion.
Det er vanlig å finne mikrosatellitter inne i de ikke -kodende haplotypene, derfor kan forskjellige alleler observeres i antall repetisjoner av mikrosatellitter, og gjennom påvisning av variasjoner i antall repetisjoner av mikrosatellitter.
Mikrosatellittmolekylære markører er utviklet for påvisning av uendelige haplotyper, fra sexat av planter som papaya (Carica Papaya) til påvisning av menneskelige sykdommer som falciform anemi.
Polymorfismer av de amplifiserte fragmentene (AFLP)
Denne teknikken kombinerer amplifisering med PCR -reaksjoner med DNA -fordøyelse med to forskjellige restriksjonsenzymer. Teknikken oppdager polymorfe loci i haplootyper i henhold til de forskjellige skjærestedene i DNA -sekvensen.
For å illustrere teknikken bedre, la oss forestille oss tre stofffragmenter av samme lengde, men kuttet på forskjellige steder (disse fragmentene representerer tre fragmenter av amplifiserte haplotyper ved bruk av PCR -teknikken).
På det tidspunktet stoffet er kuttet, vil mange deler av forskjellig størrelse oppnås, siden hver klut kuttes på forskjellige steder. Når vi bestiller fragmentene i henhold til typen stoff de kommer fra, kan vi observere hvor forskjellene mellom stoffer eller haplotyper blir funnet.
Diagnostikk og sykdommer
En viktig fordel med den genetiske studien av haplotyper er at disse forblir nesten intakte eller uendret i tusenvis av generasjoner, og dette tillater identifisering av eksterne forfedre og hver av mutasjonene som individer bidrar til utvikling av sykdommer.
Kan tjene deg: fenotypiske variasjonerHaplootyper i menneskeheten varierer avhengig av raser, og med utgangspunkt i dette har gener blitt påvist i haplotyper som forårsaker alvorlige sykdommer i hver av menneskeløpene.
I prosjektet Hapmap Fire rasegrupper er inkludert: europeisk, nigeriansk yoruba, kinesisk Han og japansk.
På denne måten prosjektet Hapmap Det kan dekke de forskjellige gruppene av populasjoner og spore opprinnelsen og utviklingen av mange av de arvelige sykdommene som påvirker hvert av de fire løpene.
En av sykdommene som oftest er diagnostisert ved bruk av haplotypeanalyse er falciform anemi hos mennesker. Denne sykdommen diagnostiseres ved å spore frekvensen av afrikanske haplotyper i en populasjon.
Å være en sykdom opprinnelig fra Afrika, og identifisere afrikanske haplotyper i populasjoner lar deg enkelt spore mennesker som har mutasjonen i den genetiske sekvensen for beta -globinas av erytrocytter i form av en HOZ (karakteristikk av patologi).
Eksempler
Med haplotyper fylogenetiske trær som representerer evolusjonsforhold mellom hver av haplotypene som finnes i en prøve av homologe DNA -molekyler eller samme art, i en region som har lite eller ingen rekombinasjon er konstruert.
En av de mest studerte grenene gjennom haplotyper er utviklingen av menneskers immunforsvar. Haplootyper som koder for bompengeseptoren (en nøkkelkomponent i det medfødte immunforsvaret) er blitt identifisert for neandertalen og Denisovanos -genomet.
Dette lar dem spore hvordan genetiske sekvenser har endret seg i populasjonene av "moderne" mennesker med hensyn til haplotypesekvensene som tilsvarer "forfedres" mennesker.
Å bygge et nettverk av genetiske forhold fra mitokondriell haplotype.
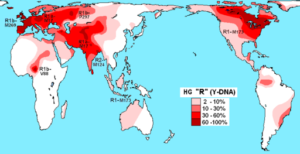 Haplogruppefordeling R (DNA-Y) i innfødte populasjoner (kilde: Maucioni [CC av 3.0 (https: // creativecommons.Org/lisenser/av/3.0)], via Wikimedia Commons) Mangfoldet av haplotyper brukes til å spore og studere det genetiske mangfoldet av dyr som er oppdrettet i fangenskap. Disse teknikkene brukes spesielt for vanskelige arter for å overvåke dyrelivet.
Haplogruppefordeling R (DNA-Y) i innfødte populasjoner (kilde: Maucioni [CC av 3.0 (https: // creativecommons.Org/lisenser/av/3.0)], via Wikimedia Commons) Mangfoldet av haplotyper brukes til å spore og studere det genetiske mangfoldet av dyr som er oppdrettet i fangenskap. Disse teknikkene brukes spesielt for vanskelige arter for å overvåke dyrelivet.
Dyrearter som haier, fugler og store pattedyr som jaguarer, elefanter, blant andre, blir kontinuerlig genetisk evaluert gjennom mitokondrielle haplotyper for å overvåke den genetiske tilstanden til populasjoner i fangenskap.
Referanser
- Bahlo, m., Stankovich, J., Hastighet, t. P., Rubio, J. P., Burfoot, r. K., & Foot, S. J. (2006). Deteting genom bred haplotypedeling ved bruk av SNP eller mikrosatellitt haplotypedata. Human Genetics, 119 (1-2), 38-50.
- Dannemann, m., Andrés, a. M., & Kelso, J. (2016). Introgress av neandertaler og denisovanske lignende haplotyper bidrar til adaptiv variasjon i menneskelige bompenger-lignende mottakere. The American Journal of Human Genetics, 98 (1), 22-33.
- Fra vries, h. G., Van der Meulen, m. TIL., Rozen, r., Halley, d. J., Scheffer, h., Leo, s.,... og du meerman, g. J. (nitten nittiseks). Haplotype identitet mellom individer som deler en CFTR -mutasjonsallel. Human Genetics, 98 (3), 304-309
- Swallow, m. TIL., Leaver, a. L., Christiansen, f. T., Witt, c. S., Abraham, l. J., & Dawkins, r. L. (1992). Forfedres haplotyper: Conserva Population MHC haplotyper. Menneskelig immunologi, 34 (4), 242-252.
- Fellows, m. R., Hartman, t., Hermelin, d., Landau, g. M., Rosamond, f., & Rozenberg, L. (2009, juni). Haplotype inferens begrenset av plausible haplotypedata. I årlig symposium om samsvarende kombinatorisk mønster (PP. 339-352). Springer, Berlin, Heidelberg.
- Gabriel, s. B., Schaffner, s. F., Nguyen, h., Moore, J. M., Roy, J., Blumenstiel, f.,... & liu-cordero, s. N. (2002). Strukturen til haplotypeblokker i det menneskelige genomet. Science, 296 (5576), 2225-2229.
- International HapMap Consortium. (2005). Et haplotypekart over det menneskelige genomet. Nature, 437 (7063), 1299.
- Wynne, r., & Wilding, C. (2018). Mithochondrial DNA Haplotype Diversity and Origin of Captive Sand Tiger Sharks (Carcharias Taurus). Journal of Zoo and Aquarium Research, 6 (3), 74-78.
- Yoo, og. J., Tang, J., Kaslow, r. TIL., & Zhang, k. (2007). Haplotype inferens for nåværende genotypedata ved bruk av previusly identifiserte haplotyper og haplotypemønstre. Bioinformatikk, 23 (18), 2399-2406.
- Young, n. S. (2018). Aplastisk anemi. New England Journal of Medicine, 379 (17), 1643-1656.
- « Hollandiske arvegenskaper, gener av gener, degenerasjon
- Lentén større egenskaper, habitat, egenskaper, omsorg »

